笔记本实验
本笔记本基于Amazon Braket实现了Quantum Speedup for Protein Structure Prediction。这主要由Renata Wong 和 Weng-Long Chang贡献。
使用Grover算法的蛋白质折叠
蛋白质折叠是蛋白质分子呈现其三维形状的过程, 这对其正常功能至关重要。 蛋白质由氨基酸的线性链组成, 它们的最终结构由氨基酸的序列和它们之间的相互作用决定。
在蛋白质折叠过程中,线性氨基酸链折叠成独特的三维结构,通过各种类型的相互作用(例如氢键、静电力和范德华力)来稳定该结构。蛋白质折叠的过程非常复杂,涉及多个阶段,包括二级结构的形成,例如 alpha 螺旋和 beta 折叠,以及将这些结构包装成最终的三维形状。
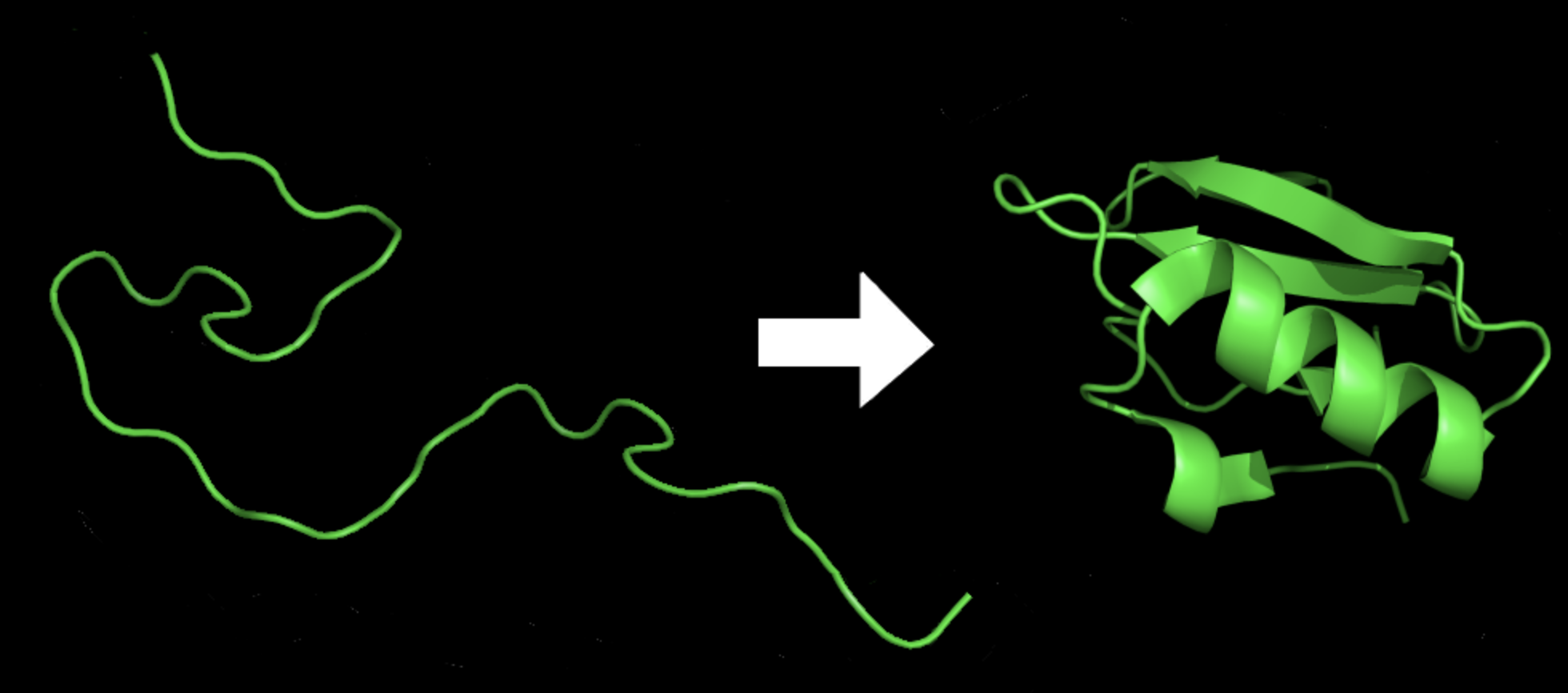
图11: 折叠前后的蛋白质8
在这项工作中,基于快速量子算法 建议使用 Grover 搜索。蛋白质结构 预测问题在研究 体心立方晶格的三维疏水-亲水模型。 结果显示二次加速 超过其经典同行。
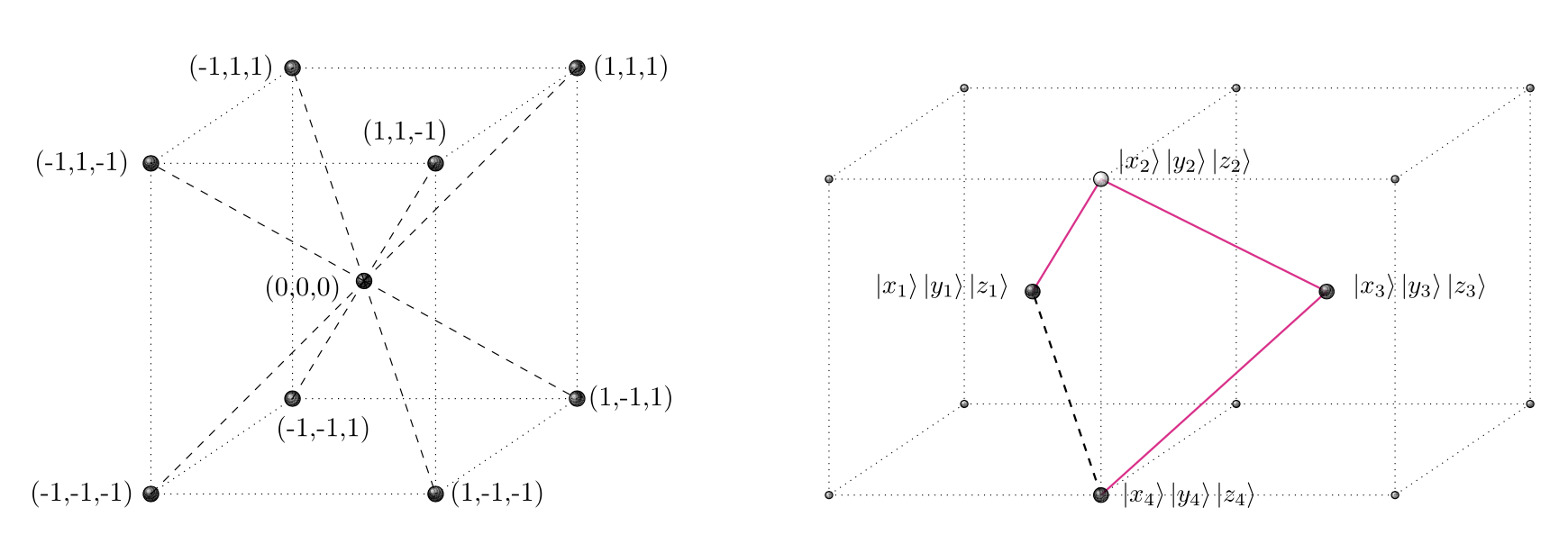
图12: 体心立方晶格[11]
Grover 算法是一种量子算法,它 可用于搜索未分类的数据库 O(sqrt(N)) 时间内的 N 项。这是一个 显着加速相比 经典算法,需要 O(N) 时间来搜索未排序的数据库。
部署完成后,您可以在堆栈页面选择解决方案的根堆栈,选择输出(Outputs),打开笔记本的链接。请到healthcare-and-life-sciences/c-3-protein-folding-grover-search/protein-folding-gs.ipynb查看细节。